バラ科果樹の系統進化学的研究
ナシ、リンゴ、モモ、スモモ、アンズ、ウメ、ビワなどの果物はバラ科に属しています。これらはバラの遠い親戚にあたります。私たちは現在これらの果物の起源や類縁関係を葉緑体DNAを使って調べています。バラ科果樹の類縁関係を明らかにすることによって進化の道筋を知り、また遺伝育種学的な現象の解明に役立てていこうとしています。現在系統関係を決めるために葉緑体DNAのどの部分を使えば有効か調べています。例えばナシ属とサクラ属の葉緑体DNAの制限酵素地図を作製しました(Katayama
& Uematsu, 2002; Katayama & Uematsu 2003)。また次世代シーケンサーを用いてニホンナシ’豊水’葉緑体ゲノムの全塩基配列を決定し、リンゴとの構造比較からゲノム中のDNA断片の欠失や挿入、転座がみつかり、また葉緑体SSR配列を同定しました(Terakami
et al. 2012)。これらの情報を使ってナシ属やサクラ属の進化の道筋を明らかにする事を目指しています。
ニホンナシ’豊水’葉緑体ゲノムの遺伝子地図
バラ科4亜科植物の葉緑体ゲノムContig Phage clone library
バラ科4亜科の葉緑体ゲノムの構造解析を行うために葉緑体ゲノムの全領域をカバーする並列ファージクローンライブラリーを作製しました。ナシ亜科のアオナシ、サクラ亜科のモモ、シモツケ亜科のトサシモツケ、バラ亜科のイチゴの葉緑体ゲノム全域がクローニング出来ました。現在これらのクローンを用いて葉緑体ゲノムの詳細な構造解析とゲノム構造の比較を進めています。
サクラ属植物の葉緑体ゲノムの構造解析(進化、遺伝的多様性、不連続変異)
サクラ属の葉緑体ゲノムの構造解析はこれまでほとんど行われていませんでした。私たちのグループはサクラ属の葉緑体ゲノム構造を把握することと、モモの進化の道筋を理解することを目的として中国のモモを用いて葉緑体ゲノムの構造解析を行っています。これまでにモモの葉緑体ゲノムの制限酵素地図を作製しました。またサクラ属22種を用いて5種類の制限酵素によりRFLP分析を行い多型もマッピングしました。その結果、サクラ属の葉緑体ゲノムは13種類のゲノムタイプが見つかり、ゲノム上に突然変異が集中して起こる領域があることが明らかになりました。現在なぜ変異の集中領域が生じるのか塩基配列上の特徴を調べています。また中国大陸由来のモモに種特異的な構造変異も見
いだしており系統進化学的な研究の指標として利用しています(Katayama & Uematsu 2005,TAG)。
ナシ属葉緑体ゲノムの構造解析(高い保存性、超可変領域の構造、進化)
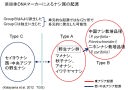
ナシの起源を推定するため世界のナシ属植物23種を用いて系統進化学的研究を行っています。ヨーロッパ、中央アジア、西アジア、ロシア、インド、アフリカ、中国、日本由来のナシの進化の道筋を葉緑体DNAマーカーを用いて推定しています。これまでの結果、西洋ナシ、西アジア、中央アジア、ロシア、アフリカの野生梨グループと東アジアの砂梨、ニホンナシ栽培種、白梨、インドナシのグループ、東アジアのマメナシ、ホクヤマナシ栽培品種、アオナシ、イワテヤマナシの3つのグループに分けることが出来ました。その結果、ナシ属が多起源であることを推定しました。(Katayama
et al. 2007, Katayama et al. 2012)。
参考文献
Nashima, K., Terakami, S., Nishitani, C., Kunihisa, M., Shoda, M., Takeuchi,
M., Urasaki, N., Tarora, K., Yamamoto, T., Katayama, H. (2015): Complete
chloroplast genome sequence of pineapple (Ananas comosus). Tree Genetics & Genomes 11, 60 doi10.1007/s11295-015-0892-8.:
Katayama, H., Tachibana M., Iketani, H., Zhang, Shao-ling, Uematsu, C.
(2012) Phylogenetic utility of structural alterations found in the chloroplast
genome of pear: Hypervariable regions in a highly conserved genome. Tree
Genetics and Genomes vol8 313-326.
Terakami, S., Matsumura Y., Kurita, K., Kanamori, H., Katayose, Y., Yamamoto,
T., Katayama, H. (2012) Complete sequence of the chloroplast genome from
pear (Pyrus pyrifolia): genome structure and comparative analysis. Tree
Genetics and Genomes 8,841-854
.
Katayama H., Adachi S., Yamamoto T., Uematsu C.: A wide range of genetic
diversity in pear genetic resources from Iwate, Japan revealed by SSR and
chloroplast DNA markers,Genetic Resources and Crop evolution 2007, 54:1573-1585
Katayama H. & Uematsu C.: Pear (Pyrus species) genetic resources in Iwate, Japan, Genetic Resources and Crop evolution,2006, vol.53, 483-498
Katayama H. & Uematsu C.: Structural analysis of chloroplast DNA in
Prunus (Rosaceae): evolution, genetic diversity and unequal mutations,
Theor Appl Genet, 2005, vol 111, 1430-1439
Katayama H. & Uematsu C.: Comparative analysis of chloroplast DNA in
Pyrus species: physical map and gene localization, Theor Appl Genet 2003 vol.106,303-310
Katayama H. & Uematsu C.: Phylogenetic Analysis of Pyrus species:The
chloroplast DNA structure of Pyrus ussuriensis var. hondoensis, Acta Hort; 2002 vol.587, 259-268
学術講演会(招待講演含む)
奈島賢児・寺上伸吾・ 西谷千佳子・國久美由紀・正田守幸・竹内誠人・浦崎直也・太郎良和彦・山本俊哉・片山寛則(2014)パインアップルのクロロプラストゲノムDNAの全長配列 の決定とその解析,園芸学研究第13巻別2,p111.
片山寛則(2012) 次世代シーケンサーを用いたナシ葉緑体DNAの全塩基配列決定,園芸学平成24年春季大会,小集会;‘ナシゲノム研究の発展と展望’
松村有一郎・寺上伸吾・栗田加奈子・金森裕之・片寄裕一・山本俊哉・片山寛則(2011)ニホンナシ’豊水’の葉緑体ゲノムの全塩基配列決定、育種学研究第13巻別1,p287.
片山寛則 2006 バラ科果樹における葉緑体ゲノムの進化
(園芸学会秋季大会 シンポジウム バラ科果樹におけるゲノム研究の進展と展望にて)
なおこれらの分子系統学的な研究は大阪市立大学理学部附属植物園、果樹研究所との共同研究です。